Allgemeines Projekt
Deep Glant Hogweed: Automatisierte Erkennung von Neophyten mittels multispektralen Luftbildern
Invasive Pflanzen, sogenannte Neophyten, stellen Kommunen und Gemeinden vor grosse Herausforderungen. Neophyten bedrohen naturbelassene Gebiete, verdrängen einheimische Arten und können im Extremfall sogar gesundheitsschädlich sein (z.B. Riesenbärenklau, Heracleum mantegazzianum). Ein verlässliches Monitoring ist Grundvoraussetzung für die Bekämpfung dieser Pflanzen. Mittels Drohnentechnologie können automatisiert multispektrale Luftaufnahmen der betroffenen Gebiete erfasst und mittels Methoden des Maschinellen Lernens mit hoher Zuverlässigkeit erkannt und klassifiziert werden. Um die Strukturen in der Vegetation hervorzuheben, werden relevante, aus der Literatur bekannte Vegetationsindizes angewendet. Diese Vegetationsindizes dienen als Merkmale für die Machine Learning Klassifikatoren. Um die Robustheit zu erhöhen, wird die Klassifikation nicht auf Pixelebene, sondern auf sogenannten Superpixeln ausgeführt. In dieser Arbeit konnte die Praxistauglichkeit des Ansatzes anhand eines einfachen Benchmark Szenarios nachgewiesen werden.
Neophyten erkennen
Neophyten sind gebietsfremde Pflanzen, welche absichtlich oder versehentlich in die örtliche Flora eingeführt wurden und in Folge verwildert sind. Dabei wird zwischen invasiven Neophyten und Neophyten mit invasivem Potenzial unterschieden. Erstere dürfen weder in Verkehr gebracht, noch importiert, verkauft, transportiert oder gepflanzt werden. Sie charakterisieren sich durch eine starke Ausbreitung und verdrängen häufig die einheimische Flora. Gewisse invasive Neophyten sind gesundheitsschädigend, andere destabilisieren beispielsweise Bachufer oder schädigen Bauten. Aktuell gelten in der Schweiz 41 Pflanzenarten als invasive Neophyten und 17 als potenziell invasiv. Letztere, die Neophyten mit invasivem Potenzial, sind informationspflichtig und beim Umgang mit diesen dürfen weder Menschen, Tiere noch die Umwelt gefährdert oder die Biodiversität beeinträchtigt werden bei deren Vertrieb. [1].
Neophyten wie Silybum marianum (Mariendistel) und andere invasive Unkräuter stellen eine bedeutende Herausforderung für die landwirtschaftliche Produktivität dar, da sie wertvolle Ressourcen wie Nährstoffe und Wasser beanspruchen. Die traditionelle Unkrautbekämpfung, z. B. durch chemische Herbizide oder manuelle Entfernung, ist ineffizient und schädlich für die Umwelt. UAVs bieten eine effiziente Möglichkeit, grosse landwirtschaftliche Flächen zu überwachen und durch multispektrale Bildgebung genaue Daten zu erfassen, die zur Unterscheidung zwischen Unkraut und Nutzpflanzen verwendet werden können.
Merkmalsextraktion über Vegetationsindizes und Superpixel
In diesem Projekt wurde mit klassischer Merkmalsextration und klassischen ML-Algorithmen versucht, Neophyten zuverlässig zu erkennen. Dafür wurden spektrale Eigenschaften (Vegetationsindizes) sowie strukturelle Eigenschaften der Neophyten (Superpixel) als Merkmale verwendet.
Vegetationsindizes machen sich den starken Anstieg des Reflexionsgrades photosyntheseaktiver Vegetation vom roten (ca. 630–690 nm) zum nah-infraroten (ca. 750–900 nm) Bereich zunutze. Chlorophyll, besser: Mesophyll, reflektiert im nahen Infrarot ungefähr sechsmal stärker als im sichtbaren Spektrum. Zu den gemessenen Vegetationsmerkmalen gehören unter anderem der Blattflächenindex (LAI), der Prozentsatz der grünen Bedeckung, der Chlorophyllgehalt, die grüne Biomasse und die absorbierte photosynthetisch aktive Strahlung (APAR). Diese Vegetationsindices werden für jedes Pixel der Luftaufnahmen berechnet und dienen als Merkmale für die Klassifikations-Algorithmen.
Superpixel liefern strukturelle Merkmale der Vegetation. Für die Berechnung der Superpixel wird die sogenannte SLIC Methode verwendet. Die SLIC-Methode [2] (Simple Linear Iterative Clustering) ist ein einfacher Algorithmus, der verwendet wird, um sogenannte Superpixel zu erstellen. Aber was sind Superpixel? Anstatt jedes einzelne Pixel in einem Bild separat zu betrachten, fasst man bei Superpixeln Gruppen von Pixeln zusammen, die sich ähnlich sind. Diese Gruppen stellen oft bestimmte Regionen im Bild dar, wie etwa Blätter, Äste oder Boden, und bieten eine vereinfachte Darstellung der Bildinhalte. Um diese Superpixel zu erzeugen, nutzt der SLIC-Algorithmus einen Prozess, der ähnlich wie der k-Means-Algorithmus funktioniert, den du dir wie eine Methode zur Gruppierung vorstellen kannst. Hier sind die Hauptschritte:
- Start mit gleichmässiger Aufteilung: Zuerst werden im Bild regelmässig verteilte Punkte gewählt, die als Startpunkte für die Cluster dienen. Jedes dieser Cluster wird die Basis für einen Superpixel.
- Gruppierung ähnlicher Pixel: Der Algorithmus überprüft die Umgebung jedes Startpunkts und gruppiert die benachbarten Pixel, die ihm ähnlich sind (z.B. in Farbe und Position). So entstehen erste Cluster von Pixeln.
- Wiederholter Feinschliff: Dieser Prozess wird mehrere Male wiederholt, wobei die Pixel jedes Mal in das Cluster verschoben werden, dem sie am ähnlichsten sind. Durch diesen iterativen (schrittweisen) Prozess werden die Superpixel nach und nach besser an die Bildstruktur angepasst.
Klassifikation der Neophyten
Auf Basis dieser Merkmale, wurden gemäss dem No-Free-Lunch Theorem verschiedene klassische Modelle zur Klassifizierung für drei nicht-invasive Neophyten trainiert.
Korrektklassifizierungsrate | Neophyt 1 | Neophyt 2 | Neophyt 3 |
ML Classifier |
|
|
|
Multilayer Neural Network | 0.98 | 0.96 | 0.93 |
K-Nearest Neighbor | 0.98 | 0.95 | 0.93 |
Logistic Regression | 0.98 | 0.96 | 0.94 |
Bagging Classifier | 0.98 | 0.96 | 0.94 |
Descision Tree Classifier | 0.98 | 0.95 | 0.93 |
Random Forrest Classifier | 0.98 | 0.95 | 0.94 |
Tabelle 1: Korrektklassifizierungsrate (accuracy) auf denTestdaten von drei Neophyten für verschiedene ML-Klassifizierern.
Diese Korrektklassifizierungsraten sind sehr hoch, was darauf zurückzuführen ist, dass der Trainings- und Testdatensatz aufgrund der limitieren Zeit nicht genug gross und damit divers genug war, um auf allgemeine Grünflächen zu generalisieren. Die Raten beziehen sich auf diesen limitierten Testdatensatz.
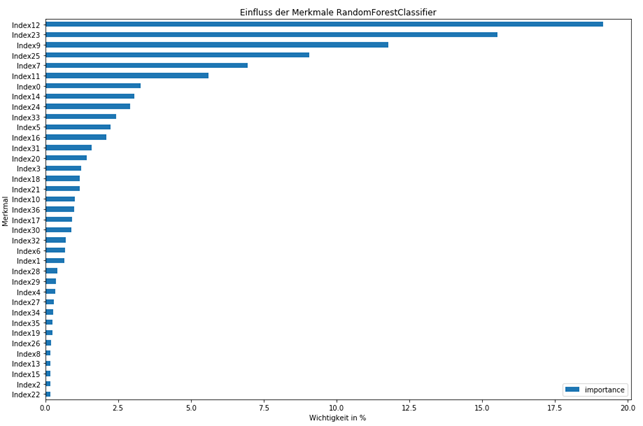
Um eine Aussage über die Wichtigkeit der Vegetationsindizes zu treffen, wird die Wichtigkeit der (permutation importance) der Indizes herangezogen. Ein Beispiel ist in der Abbildung 4 ersichtlich.
Schlussfolgerung und Ausblick: Deep-Learning Ansätze
Im Rahmen dieser Projektarbeit wurden klassische ML-Algorithmen verwendet, um drei verschiedene Neophyten zu klassifizieren. Auf dem limitierten Testdatensatz hat dies auch gut funktioniert, jedoch ist der Trainingsdatensatz nicht ausreichend, um Klassifizierer zu trainieren, welche auf allgemeinen Grünflächen bei unterschiedlichen Witterungs- und Einstrahlungs-bedingungen auch gut generalisieren. Hierfür müssten zusätzliche Drohnenflüge geplant und durchgeführt werden. Dennoch zeigen die Ansätze, dass die spektralen Merkmale durch die Vegetationsindizes als auch die strukturellen Merkmale durch die Superpixel brauchbare Merkmale liefen, um Neophyten von der restlichen Vegetation zu unterscheiden.
Stand der Technik heute ist die Verwendung von Deep-Learning Architekturen wie R-CNN, U-Net oder SegNet.
- R-CNN (Region-based Convolutional Neural Network, [3]) identifizieren Regionen im Bild, die potenziell interessante Objekte enthalten könnten. Diese Regionen werden dann durch ein CNN analysiert, um die Pflanzenart zu klassifizieren. Dies ist besonders hilfreich, um bestimmte Pflanzen, wie Neophyten, in multispektralen Luftbildern zu isolieren und zu erkennen.
- U-Net [4] ist besonders nützlich für die Segmentierung von Vegetation, da es die Struktur von Pflanzen innerhalb eines Bildes auf Pixel-Ebene erfassen kann. Die Architektur besteht aus einem Encoder-Decoder-Ansatz, bei dem der Encoder die wichtigen Merkmale extrahiert und der Decoder diese Merkmale in eine segmentierte Ausgabe transformiert, bei der jede Pixelgruppe einem bestimmten Neophyten zugeordnet wird.
- SegNet [5] ist eine Architektur für Bildsegmentierung, die für die Echtzeitanwendung optimiert ist. Wie U-Net verwendet auch SegNet einen Encoder-Decoder-Ansatz, jedoch mit einem Fokus auf Effizienz, da die Informationen aus den Pooling-Schritten genutzt werden, um die finale Segmentierung zu verfeinern.
Die Genauigkeit bei der Erkennung von Neophyten hängt stark von der genauen Beschaffenheit des Trainingsdatensatzes, den Witterungs- und Einstrahlungsbedingungen sowie der verwendeten Methode ab. CNN-basierte Modelle (insbesondere U-Net und SegNet) haben in Studien zur Pflanzenklassifikation oft eine Genauigkeit von 85% bis 95% erreicht.Dabei spielen multispektrale Daten eine Schlüsselrolle, da Neophyten oft durch Unterschiede im Spektralverhalten, z.B. im nahinfraroten Bereich, von einheimischen Pflanzen unterschieden werden können. Zu den Neophyten, die durch diese Methoden gut erkannt werden können, gehören:
- Japanischer Knöterich (Fallopia japonica)
- Kanadische Goldrute (Solidago canadensis)
- Riesen-Bärenklau (Heracleum mantegazzianum)
- Indisches Springkraut (Impatiens glandulifera)
Eine der grössten Herausforderungen bei der Detektion von Neophyten ist die spektrale Ähnlichkeit von Unkräutern und Nutzpflanzen. Dies kann zu Fehlklassifikationen führen, insbesondere bei Neophyten, die in der Nähe von Nutzpflanzen wachsen. Eine weitere Schwierigkeit besteht in der mangelnden Verfügbarkeit grosser und gut annotierter Trainingsdatensätze, die für die genaue Erkennung erforderlich sind.
Referenzen
[1] Dr. rer. nat. Erwin Jörg. Invasive Neophyten (Gebietsfremde Pflanzen, Problempflanzen) url: www.neophyt.ch/.
[2] Radhakrishna Achanta, Appu Shaji, Kevin Smith, Aurelien Lucchi, Pascal Fua, and Sabine Süsstrunk, SLIC Superpixels Compared to State-of-the-art Superpixel Methods, IEEE Transactions on Pattern Analysis and Machine Intelligence, vol. 34, num. 11, p. 2274 – 2282, May 2012.
[3] Girshick, R., Donahue, J., Darrell, T., & Malik, J. (2014). Rich feature hierarchies for accurate object detection and semantic segmentation. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition (pp. 580–587).
[4] Ronneberger O, Fischer P, Brox T (2015). "U-Net: Convolutional Networks for Biomedical Image Segmentation". https://arxiv.org/abs/1505.04597
[5] Vijay Badrinarayanan and Alex Kendall and Roberto Cipolla, A Deep Convolutional Encoder-Decoder Architecture for Image Segmentation, (2015), https://arxiv.org/abs/1511.00561
[6] Drishti Goel and Bhavya Kapur and Prem Prakash Vuppulur: Machine Learning Interventions for Weed Detection using Multispectral Imagery and Unmanned Aerial Vehicles -- A Systematic Review (2024), https://arxiv.org/abs/2408.06727
[7] Bouguettaya, A., Zarzour, H., Kechida, A. et al. Deep learning techniques to classify agricultural crops through UAV imagery: a review. Neural Comput & Applic 34, 9511–9536 (2022). https://doi.org/10.1007/s00521-022-07104-9